小麦抗条锈病基因挖掘与利用研究获进展
文章导读
为应对频繁变异的小麦条锈菌,研究员凌宏清团队与西北农林科技大学专家携手突破,首次在全球尺度上构建小麦抗条锈病“综合抗源”核心资源库,并成功绘制出首张小麦全基因组抗条锈病基因全景图。研究基于万余份小麦种质资源与近4.7万个抗病数据,定位431个关键QTL位点,揭示抗病基因在不同麦区的分布规律,同时发掘出三类高效抗性基因,其中一些在不牺牲产量的前提下,可抵御多种锈病小种。这项成果为绿色防控和精准育种提供了重要支持,推动小麦抗病育种迈入新阶段。
— 内容由好学术AI分析文章内容生成,仅供参考。
中国科学院遗传与发育生物学研究所/崖州湾国家实验室研究员凌宏清团队,联合西北农林科技大学教授康振生和韩德俊领衔的研究团队,在全球尺度上构建了小麦条锈病“综合抗源”核心资源库,解析了普通小麦条锈病抗性遗传基础,绘制出首张小麦全基因组抗条锈病基因全景图,为抗病基因合理利用与设计育种提供了理论支撑。
小麦条锈病是全球性流行真菌病害。由于条锈菌小种变异频繁,全球各地相继出现新小种,致使大部分生产使用的抗病基因丧失抗性。因此,亟需寻找新的抗病(基因)资源来提高小麦新品种抗性,并通过合理的基因布局来应对快速变异的条锈菌。
该研究在世界范围内征集了超过14000份普通小麦,包含国内外农家种、主栽品种、高代品系和核心种质等,根据来源、生长习性和多样性,并结合条锈病各流行区特点,挑选2191份作为多样性代表进行全基因组重测序,绘制了高密度基因组变异图谱。研究结合多年多点的近47000个条锈病抗性表型数据,利用全基因组关联荟萃分析,揭示了全球各个麦区抗条锈病的遗传基础,绘制了包含431个QTL位点的小麦基因组抗条锈病基因全景图,明确了抗病基因在各个锈病流行区的分布和利用规律,阐明了一个世纪以来小麦条锈病抗性基因选择特征和流行病学特征之间的关系。
在此基础上,结合转录表达、单倍型变异、候选基因关联分析等,该研究筛选获得559个与抗条锈病相关的候选基因,并初步对其中的三个基因进行图位克隆、突变体分析、转基因等功能验证。研究发现,这些基因总体上可分为三种类型。一是新优异等位基因Yr5x可抵抗当前潜在流行的条锈菌新生理小种;二是兼抗小麦条锈病与白粉病等位基因Yr6/Pm5,其可能通过I366T和M1011I两个关键氨基酸位点实现病原菌特异性识别;三是新型兼抗条锈病和叶锈病基因YrKB,其几乎无产量负效应。
上述研究为提高小麦抗病性提供了理论依据和优质基因(组合),有望推动普通小麦抗病分子育种进程,为实现条锈病的绿色防控奠定基础。
7月22日,相关研究成果发表在《自然-遗传学》(Nature Genetics)上。
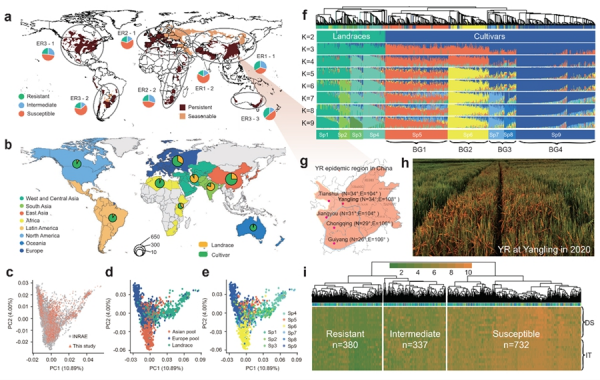
小麦条锈病流行区分布和小麦种质遗传多样性、群体结构及抗性表型多样性
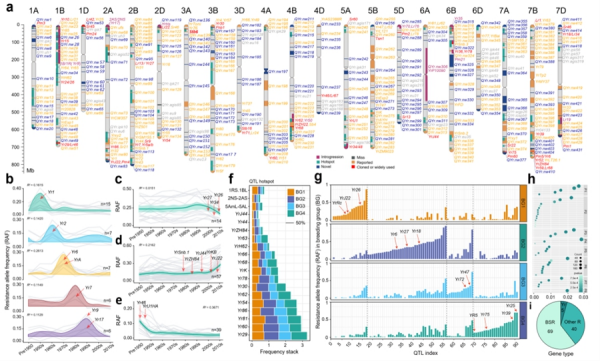
小麦抗条锈病基因全景图及抗性优异等位基因频率的时空分布
© 版权声明
本文由分享者转载或发布,内容仅供学习和交流,版权归原文作者所有。如有侵权,请留言联系更正或删除。
相关文章

暂无评论...