大模型驱动的基因组环形可视化生成研究获进展
文章导读
你是否厌倦了繁琐的脚本和僵化的工具,只为生成一张基因组可视化图?现在,科学家20分钟就能完成白血病染色体易位的交互式可视化,7分钟复现黑色素瘤突变图谱——这背后正是全新智能框架AuraGenome的突破。它用自然语言驱动大模型,将基因组数据一键转化为可交互、可追溯的环形图,效率提升69%,准确率达89%。告别代码与重复劳动,让科研真正聚焦数据洞察。这项由中国科学院团队研发的成果已发表于IEEE权威期刊,或将彻底改变基因组分析的工作范式。
— 内容由好学术AI分析文章内容生成,仅供参考。
近日,中国科学院计算机网络信息中心科研团队研发出大模型驱动的基因组环形可视化智能生成框架AuraGenome。AuraGenome突破“人工–脚本–静态”传统范式,开创“自然语言–智能体–交互”新模式,能够将基因组数据快速转化为高质量、可交互的可视化结果,并支持全流程的可追溯与复用。
在急性髓系白血病染色体易位分析中,科研人员20分钟内即可完成结构变异与转录水平的可视化生成,发现并交互式标记了潜在生物标志物区域;在黑色素瘤突变图谱复现中,研究人员仅用7分钟便高保真再现了论文中的经典可交互的可视化结果。更多对比实验表明,相比于传统工具Circos,AuraGenome效率提升69%,准确率提升至89%,实现了“让科学家专注基因组数据而非工具本身”的转变。
相关研究成果发表在《IEEE计算机图示与应用》(IEEE Computer Graphics and Applications)上。研究工作得到中国科学院战略性先导科技专项(A类)支持。
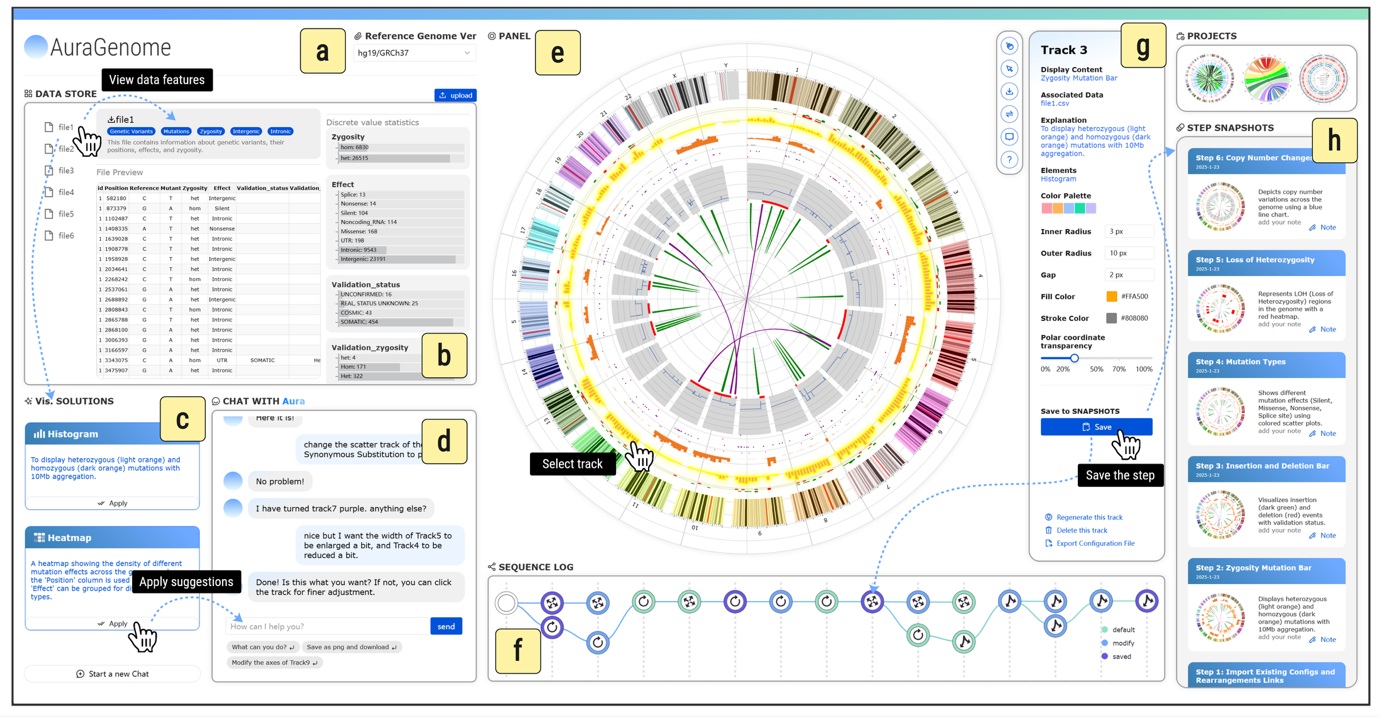
大模型驱动的基因组环形可视化自动生成可视分析系统
© 版权声明
本文由分享者转载或发布,内容仅供学习和交流,版权归原文作者所有。如有侵权,请留言联系更正或删除。
相关文章

暂无评论...