我国学者在生物正交反应驱动的蛋白质组学领域取得进展
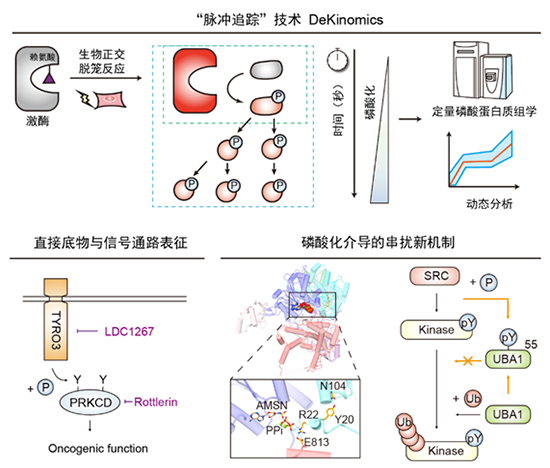 |
图 基于生物正交脱笼和磷酸蛋白质组学策略的活细胞激酶“脉冲追踪”技术DeKinomics
在国家自然科学基金项目(批准号:21937001、22137001、91753000、92253304、22125403)等资助下,北京大学陈鹏教授与南方科技大学田瑞军教授合作在磷酸蛋白质组研究领域取得新进展,相关成果以“在活细胞中使用DeKinomics对激酶功能进行脉冲-追踪研究(DeKinomics pulse-chases kinase functions in living cells)”为题,于2024年1月2日在《自然·化学生物学》(Nature Chemical Biology)上发表。论文链接:https://www.nature.com/articles/s41589-023-01497-x。
活细胞中的激酶功能研究具有很强的挑战性:一方面,在活细胞内特异追踪目标激酶的下游信号通路十分困难;另一方面,传统方式无法在高时间分辨率下发现并系统解析激酶激活的时序信息。陈鹏和田瑞军团队利用光脱笼的生物正交剪切反应实现了激酶的瞬时激活,并与定量磷酸蛋白质组学技术相结合,开发了激酶“脉冲追踪”技术(Decaging Kinase coupled proteomics,DeKinomics),实现了活细胞中特定激酶的高时间分辨组学研究,系统解析了其内源底物及下游级联信号通路。通过挖掘磷酸化蛋白质组学大数据,研究团队发现了SRC激酶对UBA1蛋白的直接磷酸化这一新通路,揭示了酪氨酸磷酸化和泛素化在修饰酶层面串扰(crosstalk)的新机制。研究团队还发现了TYRO3-PRKCD信号转导通路,提示了癌症靶向协同治疗的新思路。
该工作为研究活细胞中的激酶功能提供了普适、精准的技术,以秒级的超高时间分辨率对特定激酶的底物和下游信号通路进行系统解析。
© 版权声明
本文由分享者转载或发布,内容仅供学习和交流,版权归原文作者所有。如有侵权,请留言联系更正或删除。
相关文章

暂无评论...