中国农业大学动科学院刘剑锋教授团队提出基于多组学融合分析策略基因组选择新方法
文章导读
一项突破性方法正悄然改变基因组育种的未来。中国农业大学刘剑锋教授团队在《基因组生物学》发表全新策略FSBLUP,首次实现基因组、系谱与转录组等多组学数据的自适应融合,通过机器学习动态加权,显著提升预测准确性。在猪和小麦育种实测中,性能全面超越传统模型,尤其在低遗传力性状和数据不全场景下优势突出。该方法不仅破解了多组学整合中数据缺失与复杂耦合的难题,还具备跨物种、跨数据类型的普适性,为畜禽、作物乃至人类复杂性状的精准预测提供了可推广的创新工具。
— 内容由好学术AI分析文章内容生成,仅供参考。
近日,我校动物科学技术学院刘剑锋教授团队在国际顶级期刊《基因组生物学》(Genome Biology)在线发表研究论文《FSBLUP:一种通过优化整合中间组学数据构建融合相似性矩阵以增强基因组预测的新策略》(FSBLUP: a novel strategy of fusion similarity matrix construction via optimally integrating intermediate omics data to enhance genomic prediction),提出FSBLUP(Fusion Similarity Best Linear Unbiased Prediction)新策略,通过优化整合多组学数据构建“融合相似性矩阵”,显著提升基因组预测性能,为多组学驱动的精准育种与复杂性状遗传预测提供了新的方法工具。
基因组选择(Genomic Selection, GS)已成为动植物育种的重要技术路径。随着转录组、蛋白组、代谢组以及高通量表型等多组学数据快速积累,如何将多组学信息有效纳入预测模型,一直是本领域的研究热点与难点:不同组学层之间关联结构复杂、数据尺度差异大、缺失普遍且获取成本高;同时,传统多随机效应或多组学模型往往依赖更强的假设、需要更大的样本量,或带来更高的计算负担,导致“多组学信息真正进入育种决策”仍缺少稳定、易推广的通用工具。现有方法在数据缺失、组学层间耦合复杂或育种群体样本规模受限时,往往难以同时兼顾准确性、稳健性与可推广性。以往常见思路包括:将基因组信息与转录组/高通量表型等分别构建关系矩阵,再采用多核或多随机效应模型进行联合建模;或将高通量表型与目标性状纳入多性状框架开展联合预测等,但在真实育种场景中仍面临适用性与泛化能力不足的问题。
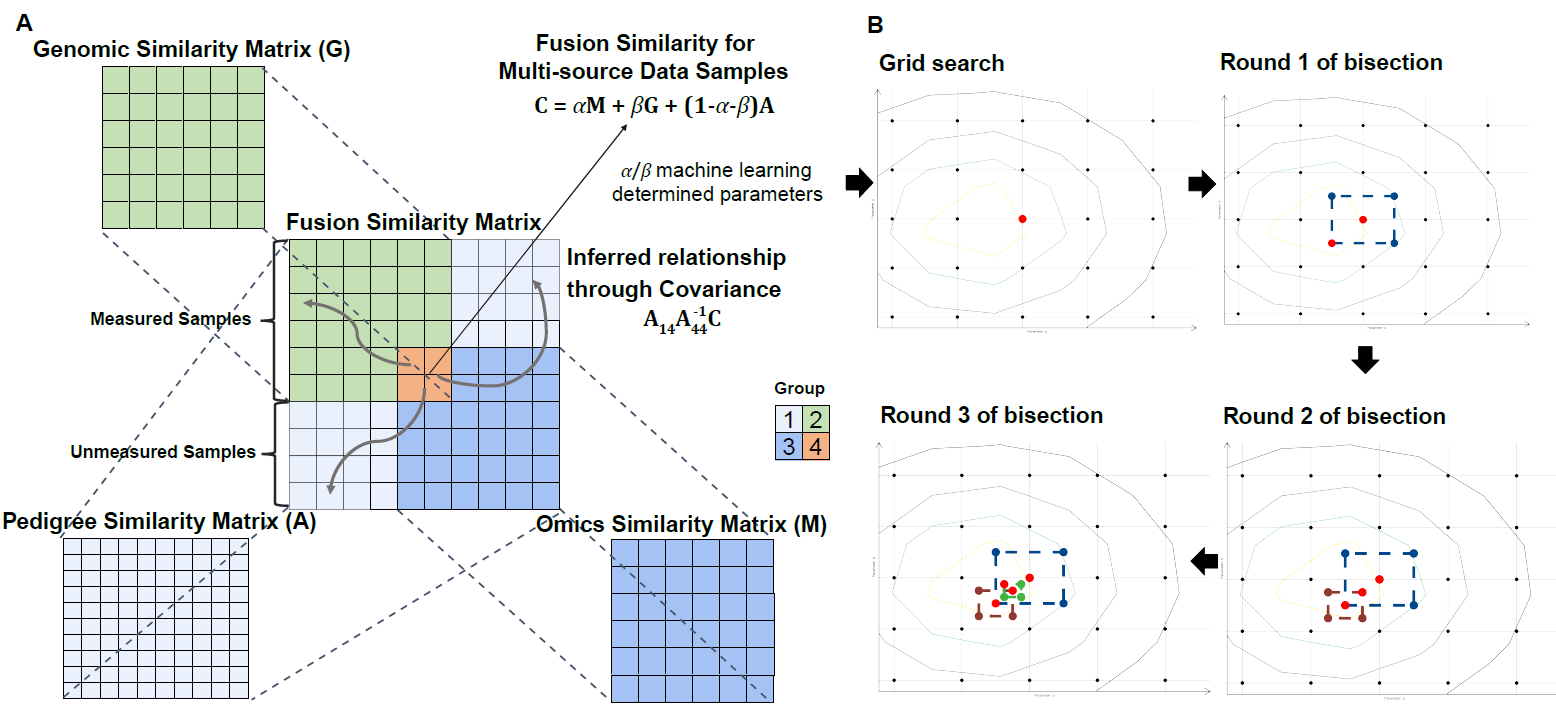
图1 FSBLUP方法框架示意图
针对上述问题,研究团队提出FSBLUP策略:在统一框架下融合基因组关系矩阵、系谱关系矩阵与中间组学(如转录组、表型组等)相似性矩阵,并通过机器学习驱动的参数优化过程(交叉验证、网格搜索与自适应二分算法等)自动估计不同数据层对表型变异的贡献,实现“按性状自适应加权”的信息整合。此外,FSBLUP还能有效应对育种实践中常见的“数据缺失”问题:对于缺少组学数据的个体,模型可利用其与已有数据个体间的系谱或基因组关系进行信息推断,尽可能确保全群体个体纳入分析,显著提升方法的可用性与推广价值。研究团队基于该模型开发了新的基因组选择软件(https://github.com/CAU-TeamLiuJF/FSBLUP),为畜禽育种分析提供了一种实用工具。
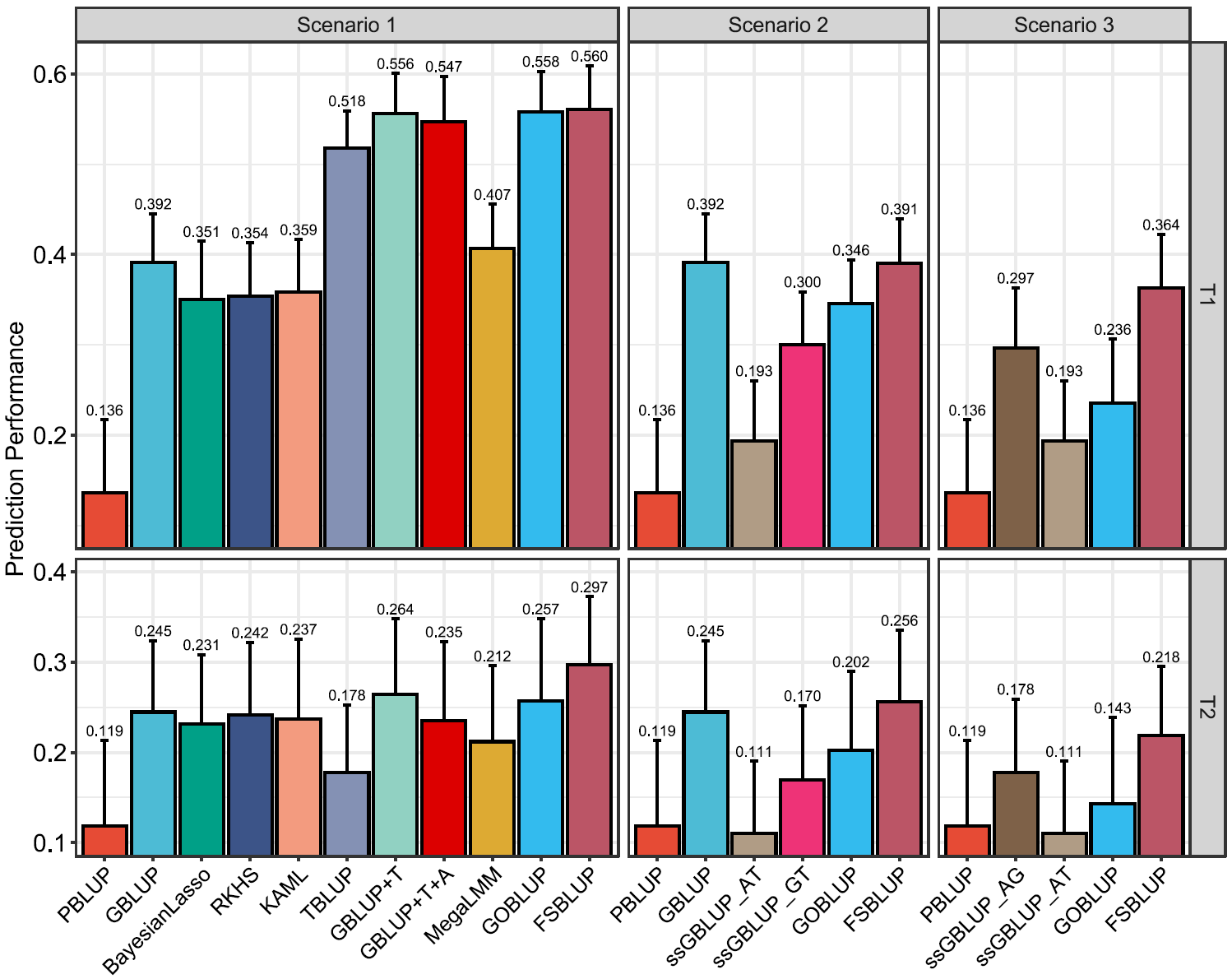
图2 FSBLUP在模拟数据中的表现
论文在模拟数据以及真实育种数据中对FSBLUP进行了系统验证。为全面评估FSBLUP性能,研究团队构建了包含高、低两类遗传力性状的模拟数据集,并设置三种数据可用性场景:(1)所有个体系谱、基因组与组学数据完整;(2)仅部分新世代个体拥有组学数据;(3)基因组和组学数据均仅在部分新世代个体中可用。结果表明,FSBLUP在多种数据可得性场景下均表现出更高或更稳定的预测准确性(图2),相较常用方法预测性能可稳定提升,尤其在多组学信息可用或性状遗传力较低等情况下优势更为明显,为多组学辅助基因组预测与精准育种提供了新的方法路径。
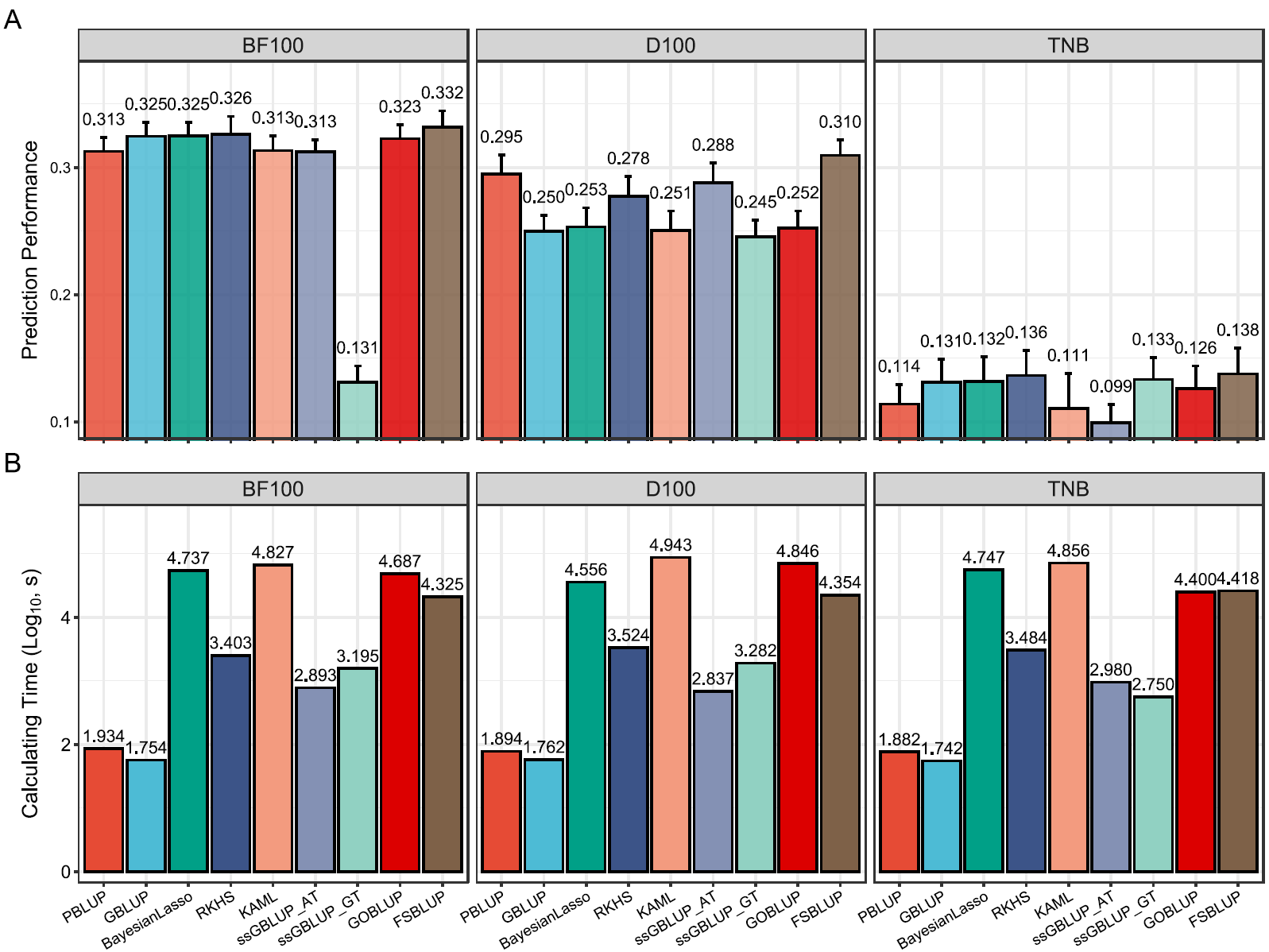
图3 FSBLUP在猪数据集中的表现
为验证FSBLUP在实际育种项目中的应用效果,研究团队将其应用于一个长白母猪育种群体。利用该群体的系谱、基因组和全血转录组数据,对达100公斤体重日龄(D100)、100公斤体重背膘厚(BF100)和总产仔数(TNB)三个关键生产性状进行预测。结果显示,FSBLUP在三个性状上的预测准确性均优于所有对比方法,平均较GBLUP和GOBLUP模型提升3%—24%(图3A)。该结果表明,FSBLUP能够从真实且可能含噪的组学数据(如全血转录组)中提取有效信息,并将其成功转化为预测准确性的提升,在畜牧育种中具有良好的应用潜力。
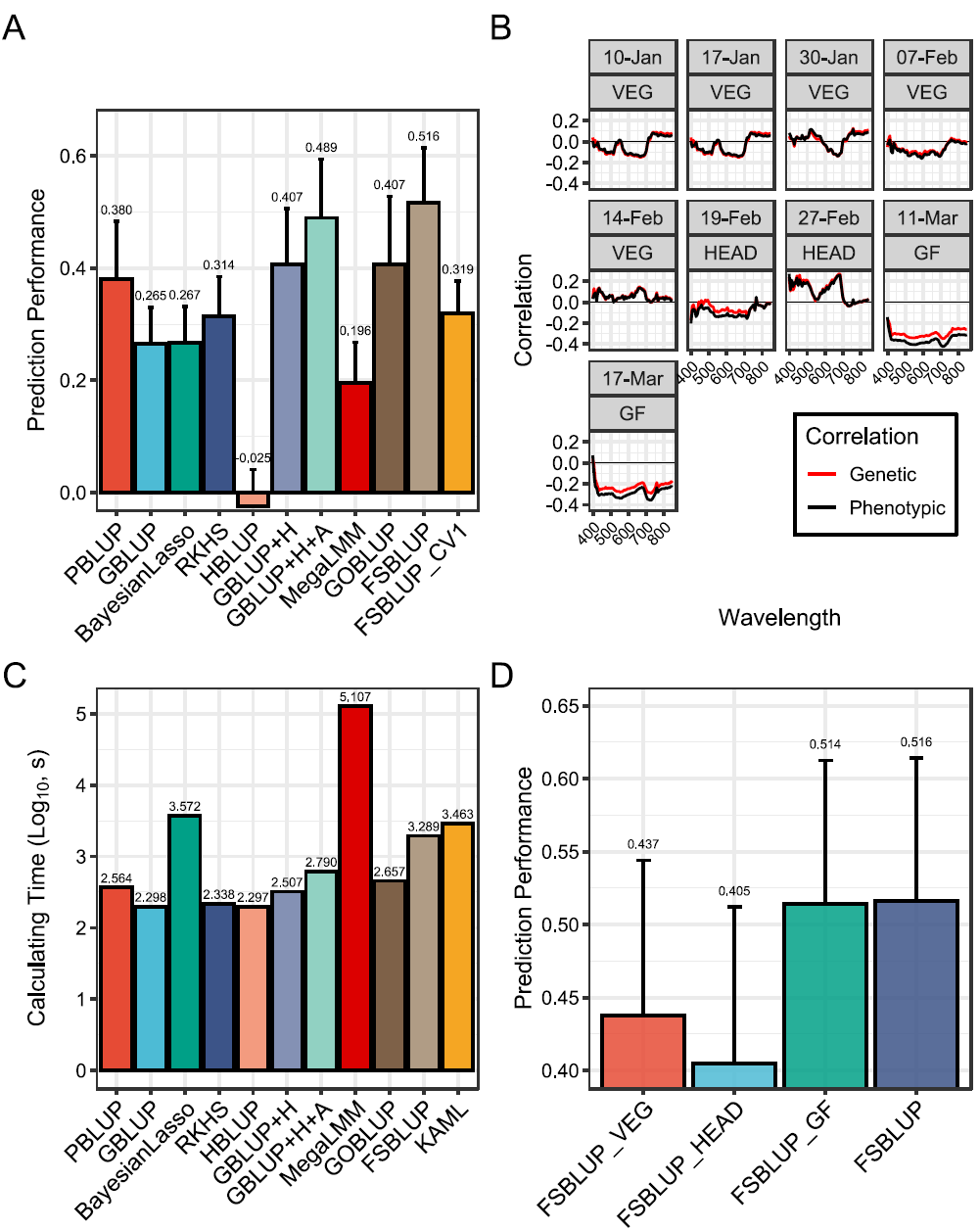
图4 FSBLUP在小麦数据集中的表现
为进一步展示FSBLUP框架的灵活性和普适性,研究团队还将其用于高通量表型辅助的作物预测场景:以小麦产量预测为例,将无人机在不同生育时期采集的高光谱反射数据视为一种特殊的“中间组学”信息。结果显示,与仅使用基因组信息的GBLUP模型(准确性0.265)相比,FSBLUP通过最优整合系谱、基因组和高光谱数据,将产量预测准确性显著提升至0.516,提升幅度达94%。FSBLUP不仅优于多种单核/多核模型,也在该数据场景下表现出对复杂多性状模型的竞争优势。其自适应加权机制能够刻画不同生育时期高光谱信息与最终产量的动态关联,从而更精准地利用表型组信息,为过程性表型驱动的精准育种提供新的方法支撑。
多组学整合预测同样是复杂疾病风险评估与遗传预测的重要方向。FSBLUP提供了一种可扩展的融合框架,有望为人类复杂性状/疾病的多组学预测研究提供新的方法选择。此外,随着微生物组、代谢组等数据在农业与生命科学领域的快速发展,FSBLUP所提供的“自适应融合—稳健预测”框架,也有望为相关方向提供更有利于精准选择与优化决策的工具支撑。
中国农业大学为论文第一完成单位,中国农业大学动物科学技术学院刘剑锋教授为通讯作者,团队博士研究生薛亚辉博士为第一作者,团队博士研究生卓越、马思佳、副教授周磊、副教授杜恒、实验师李婉影、江苏省农业科学院博士后李伟宁、北卡罗来纳州立大学助理教授蒋纪才为论文合作作者。本研究得到了科技创新-2030(2023ZD0404405)、国家自然科学基金(32272844、32302708)、中央高校基本科研业务费专项资金(2023TC196)、现代农业产业技术体系(CARS-pig-35)和中国农业大学“2115”人才培养计划等资助。本研究工作得到中国农业大学校级高性能计算平台支持。
© 版权声明
本文由分享者转载或发布,内容仅供学习和交流,版权归原文作者所有。如有侵权,请留言联系更正或删除。



















总算有能处理数据缺失的方法了,之前好多数据都白瞎了
希望多点具体的应用案例,光看数据有点抽象
看着挺厉害的,但不知道实际用起来麻不麻烦
这个软件开源了?能用在自己的数据上不?