研究实现大片段DNA染色体整合
文章导读
你是否想过,那些拯救生命的抗生素,其生产过程本身也能迎来一场“基因革命”?传统依靠不稳定质粒的生产方式,正面临效率瓶颈。现在,中国科学家取得了一项关键突破:他们开发了一种创新的基因整合工具包,能够将长达数十个基因的大片段DNA,像拼积木一样精准、稳定地“安装”到细菌染色体上。这项技术不仅成功构建出全球首株无需质粒、就能稳定合成红霉素的大肠杆菌,更将产量提升了8倍以上。这不仅仅意味着一种重要抗生素的产能飞跃,更是一把开启复杂天然产物高效生物制造新大门的通用钥匙。
— 内容由好学术AI分析文章内容生成,仅供参考。
近期,中国科学院分子植物科学卓越创新中心开发了创新的、基于可重复利用靶点的大片段DNA染色体整合策略,构建了首株能够脱离质粒、稳定合成红霉素A的大肠杆菌菌株,并提升了产量。
红霉素是临床重要的广谱大环内酯类抗生素。传统研究多采用多质粒系统进行表达,但质粒的不稳定性、抗生素的使用及细胞代谢负担过重,限制了生产效率提升。为了解决这一难题,该研究开发了基于CRISPR/Cas9的可重复利用靶点工具包。
这一工具包针对不同长度的片段采取了差异化策略。对于大于10 kb的片段,采用两步整合法,利用限制性内切酶从质粒直接切割大片段作为供体DNA,避免了长片段PCR引入突变的风险;对于小于5 kb的片段,采用迭代整合法,在同一靶点进行连续组装。通过该技术,研究将23个红霉素合成相关基因,精确集成到大肠杆菌BAP1的染色体上,获得了工程菌株sZG9,这是全球首个实现无质粒红霉素A生物合成的大肠杆菌菌株。在实现染色体稳定集成后,研究进一步针对红霉素合成中的前体供应瓶颈,进行了深度的代谢工程优化。经过多轮迭代优化,菌株sZG135的红霉素A产量达到9.80 mg/L,较初始菌株实现了8.25倍的增长,刷新了目前大肠杆菌合成红霉素A的最高产量纪录。
上述研究为红霉素的高效生产提供了高性能的底盘细胞,其开发的大片段DNA染色体整合策略具有通用性,可推广至其他复杂聚酮类、萜类等天然产物的生物合成研究,为合成生物学研究提供了工具支持。
相关研究成果在线发表在《生物资源技术》(Bioresource Technology)上。研究工作得到国家重点研发计划和国家自然科学基金等的支持。
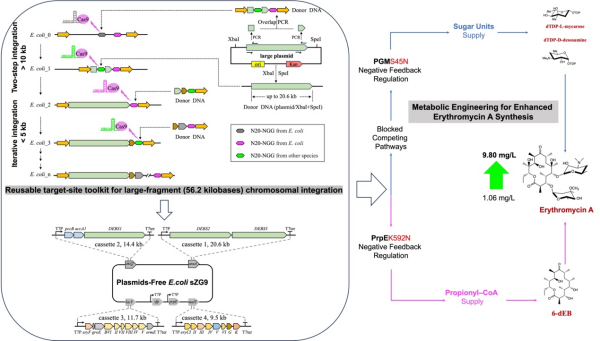
可复用靶位点工具包助力大片段DNA染色体整合并提升红霉素在大肠杆菌中的产量
© 版权声明
本文由分享者转载或发布,内容仅供学习和交流,版权归原文作者所有。如有侵权,请留言联系更正或删除。



















这玩意儿真是省事儿,直接染色体装配就能搞定,赞一个👍
产量虽然提升到9.8 mg/L,算是大幅突破,但与工业化需求相比仍有差距,后续可能要在前体供给、酶活性以及发酵工艺上多下功夫才能真正实现规模化生产。
这套工具包的可重复利用靶位点挺实用,后续如果想搬到萜类产物上,可能需要调节供体DNA的长度。
我之前在做聚酮合成时也遇到过片段不稳定的情况,换成染色体集成后效果明显提升。
大于10kb的两步整合具体操作步骤能详细点吗?
这套染色体整合方案挺牛的,省了好多质粒麻烦。