文章导读
你是否知道,95%的动物多样性竟来自无脊椎动物,而传统监测方法却难以捕捉它们的真实面貌?北京大学姚蒙团队最新研究系统评估了18对宏条形码引物,揭秘哪种引物在淡水与海洋环境中更能精准“捕获”隐秘物种。研究发现,COI引物整体表现更优,但最佳选择竟因生境而异,且数据库使用也显著影响鉴定结果。这项突破为eDNA技术标准化提供了关键指南,彻底改变水生生物多样性监测的未来格局。
— 内容由好学术AI分析文章内容生成,仅供参考。
近日,北京大学生命科学学院、生态研究中心姚蒙研究组在Methods in Ecology and Evolution发表题为“Advancing aquatic biodiversity assessments of invertebrates using eDNA metabarcoding: A systematic evaluation of primers for marine and freshwater communities”的研究论文。针对无脊椎动物多样性极高、基于宏条形码的物种鉴定方法尚缺乏统一标准的问题,该研究系统分析18对无脊椎宏条形码引物通用引物,比较它们在淡水和海洋生境中的扩增特异性、分类覆盖度与分辨率,并探讨不同序列参考数据库对物种注释的影响。该研究为水生无脊椎动物多样性调查的引物与数据库选择提供了重要信息,也为提升环境DNA(eDNA)技术在无脊椎动物监测与生态评估中的有效应用提供了重要支持。
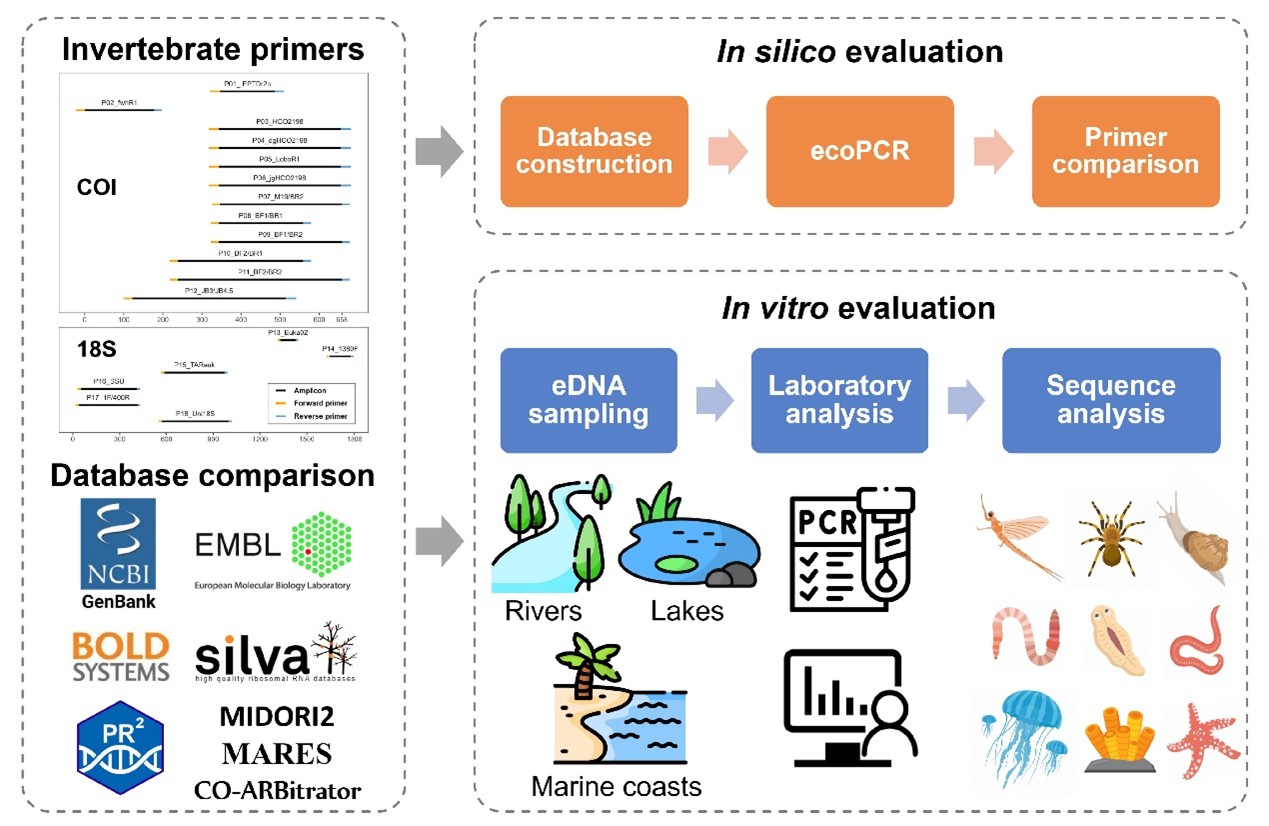
本研究的框架及主要流程
全球水生态系统面临日益严峻的生物多样性丧失,威胁生态系统稳定性及其关键服务功能。高效、精准的生物监测技术是合理有效的生态保护和管理重要前提。无脊椎动物在生态系统中发挥重要功能,也是水生生态系统健康的关键指示类群。然而,无脊椎物种多样性占全部动物的95%以上,对基于形态分类的传统调查方法造成极大挑战。eDNA宏条形码技术利用生物释放到环境中的DNA进行生物检测,具有高效、灵敏、可规模化、非侵入性的优势,已成为水生生物多样性调查的重要手段。eDNA方法的准确性高度依赖通用引物的性能。然而,目前仍缺乏针对无脊椎动物宏条形码引物的系统性比较评估,制约了该技术在无脊椎动物多样性监测中的标准化应用。
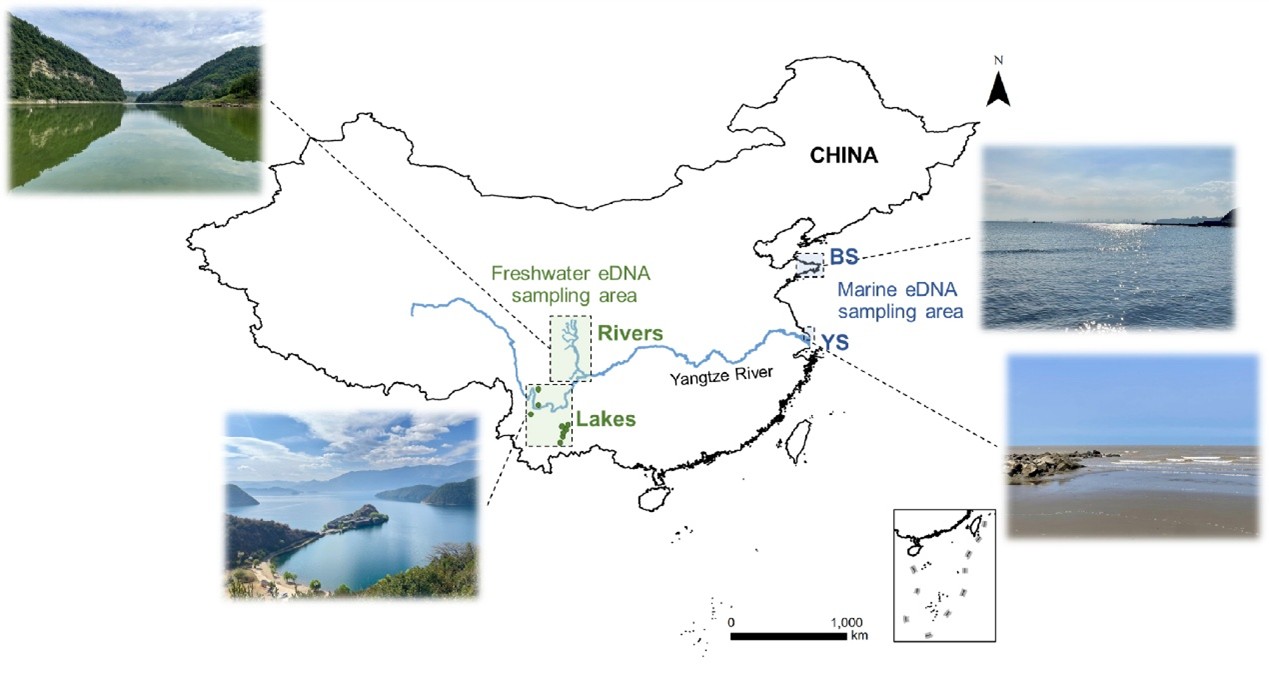
图1. eDNA采样区域图
针对这一问题,本研究利用来自中国多条河流、湖泊及沿海水体的eDNA样本(图1),通过计算机模拟(in silico)与实际PCR实验(in vitro)相结合的方法,全面评估了12对靶向线粒体COI基因和6对靶向核18S rRNA基因的无脊椎动物通用引物(图2),比较分析了这些引物在物种特异性、覆盖度、分辨率和偏好性等方面的表现。
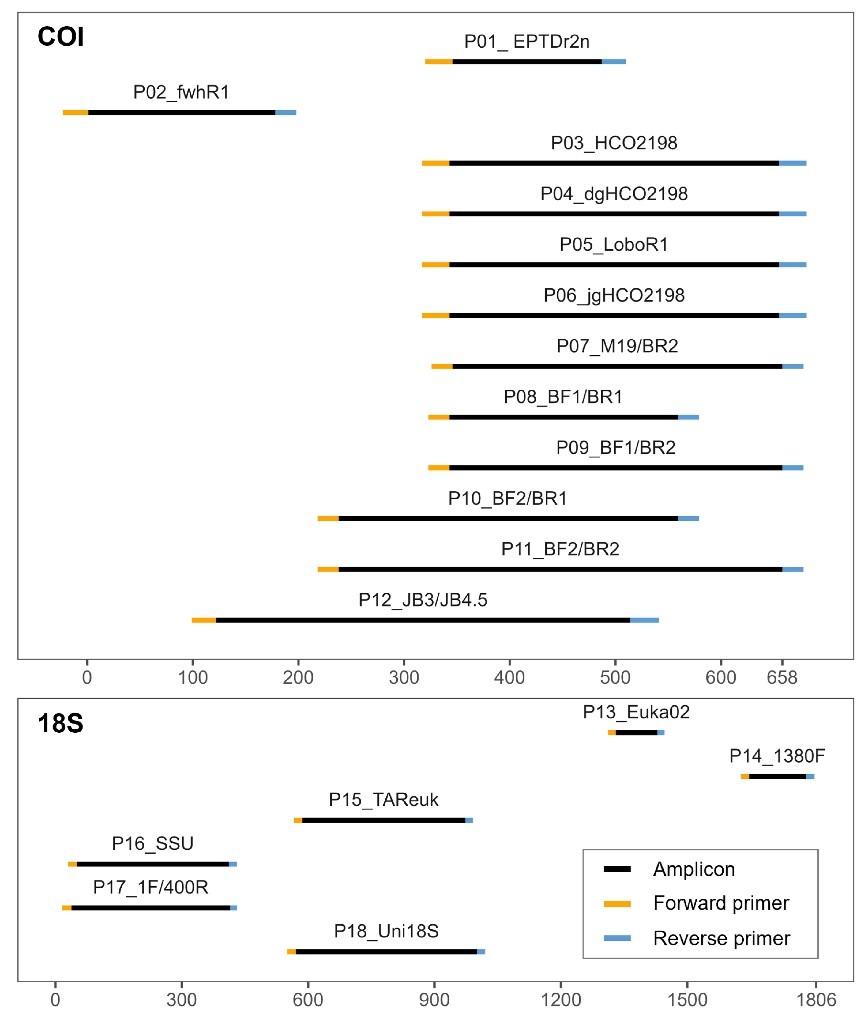
图2. 本研究中分析的无脊椎动物通用引物的靶序列位置
引物的扩增特异性在in silico和in vitro实验中均表现出显著差异(图3)。In silico结果显示,COI引物对无脊椎动物序列的扩增特异性整体优于18S引物。in vitro结果类似,但无脊椎动物序列占比相较其在in silico中的占比降低。
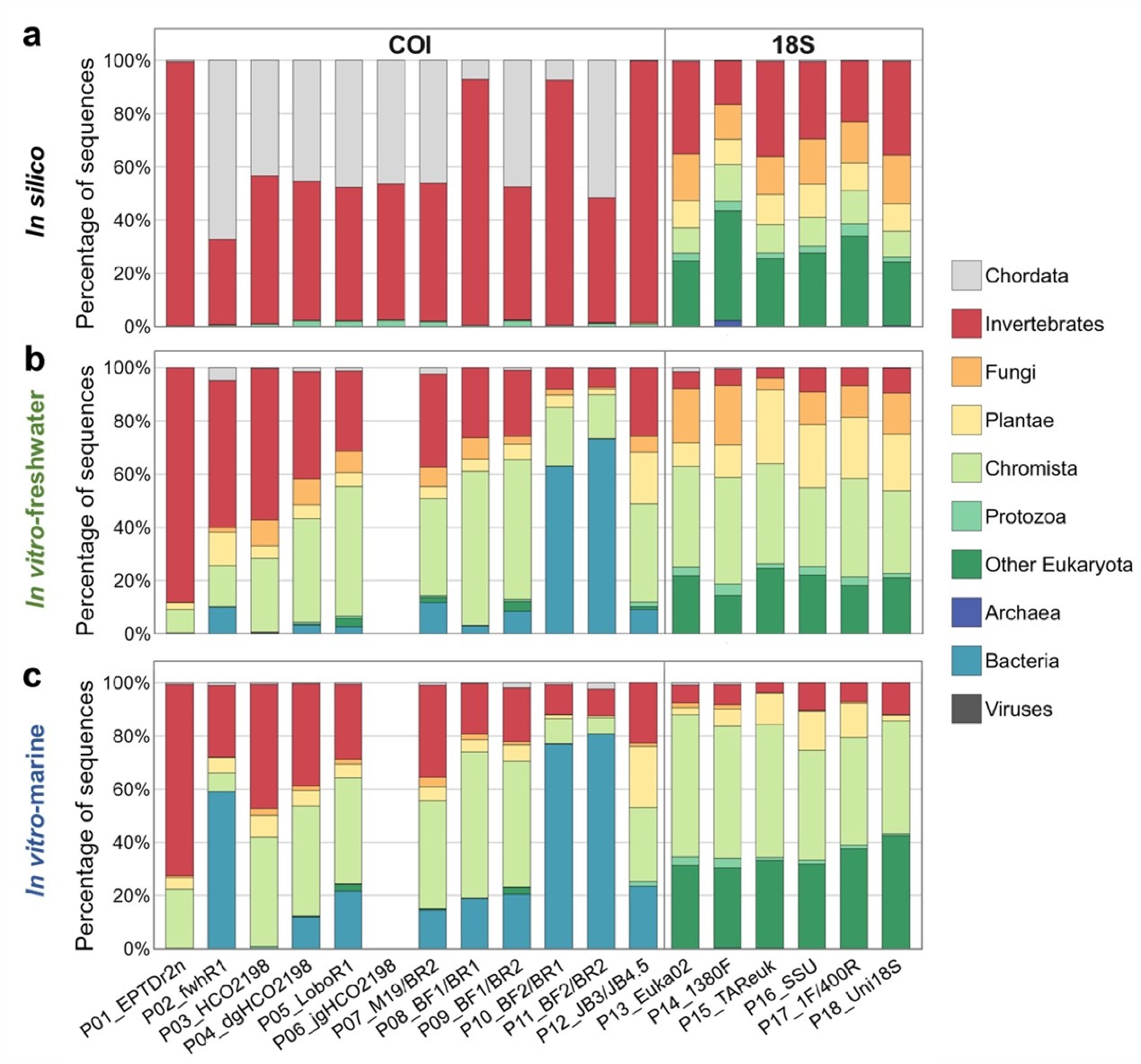
图3. COI与18S引物扩增序列的各类群组成占比。(a)基于in silico PCR分析;(b)基于淡水eDNA样本的in vitro PCR分析;(c)基于海洋eDNA样本的in vitro PCR分析
在分类覆盖度方面,COI引物检出的无脊椎动物分类单元(OTU)数量显著高于18S引物:COI引物在淡水eDNA样本中检出91—1524个无脊椎动物OTU(图4),在海洋样本中检出74—624个(图5),但分类分辨率均较低。引物P03_HCO2198和P04_dgHCO2198在淡水生境中检出的OTU数量最多,而P07_M19/BR2在海洋生境中表现最佳,显示最优引物选择受到研究环境和生物组成的影响。18S引物虽普遍检出的总OTU较少,但部分引物(如P14_1380F)能覆盖更广泛的无脊椎动物门类,可作为COI引物的有效补充。
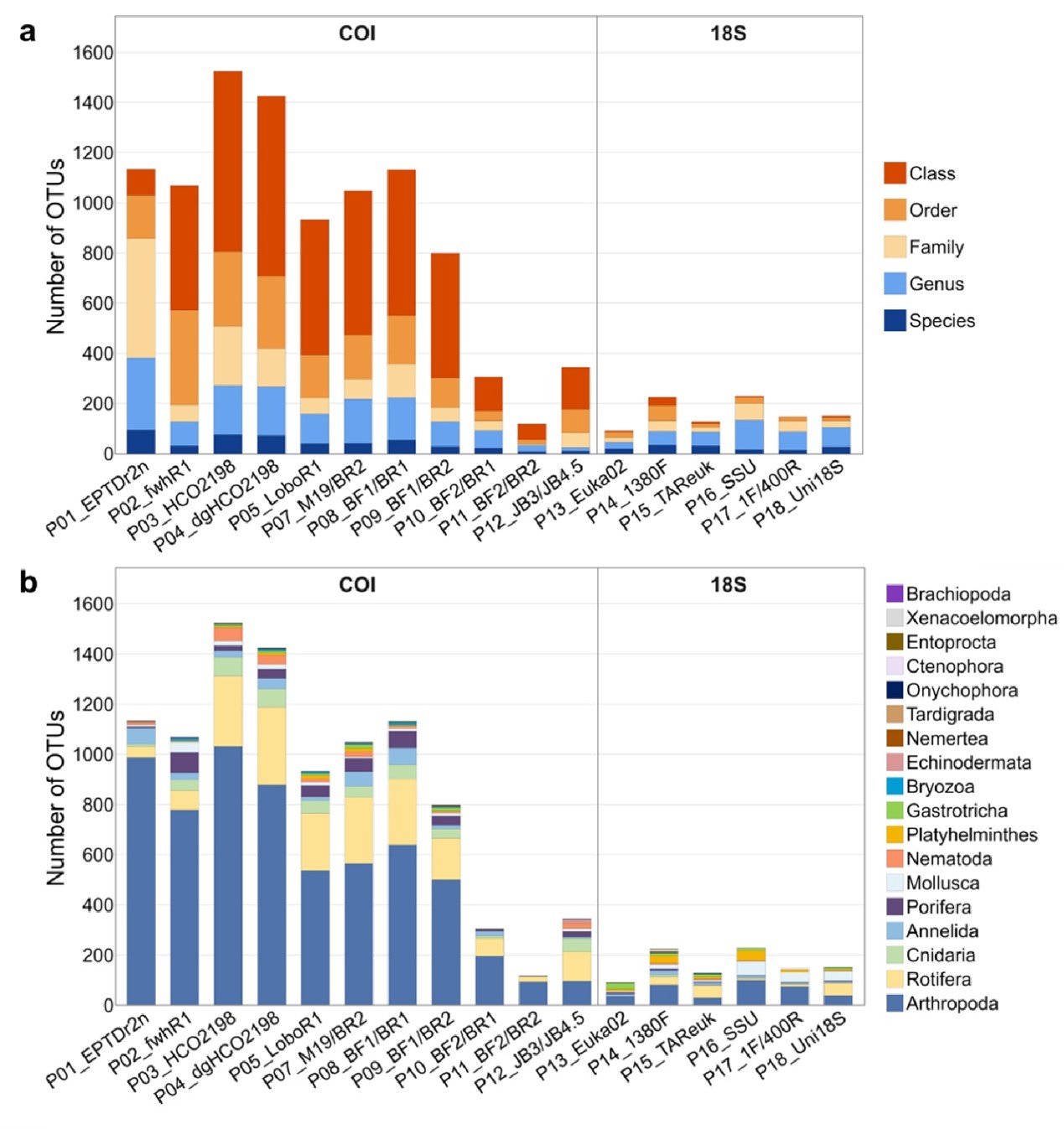
图4. 基于淡水eDNA样本的in vitro PCR分析结果,展示各引物的无脊椎分类分辨率(a)及门级类群组成(b)
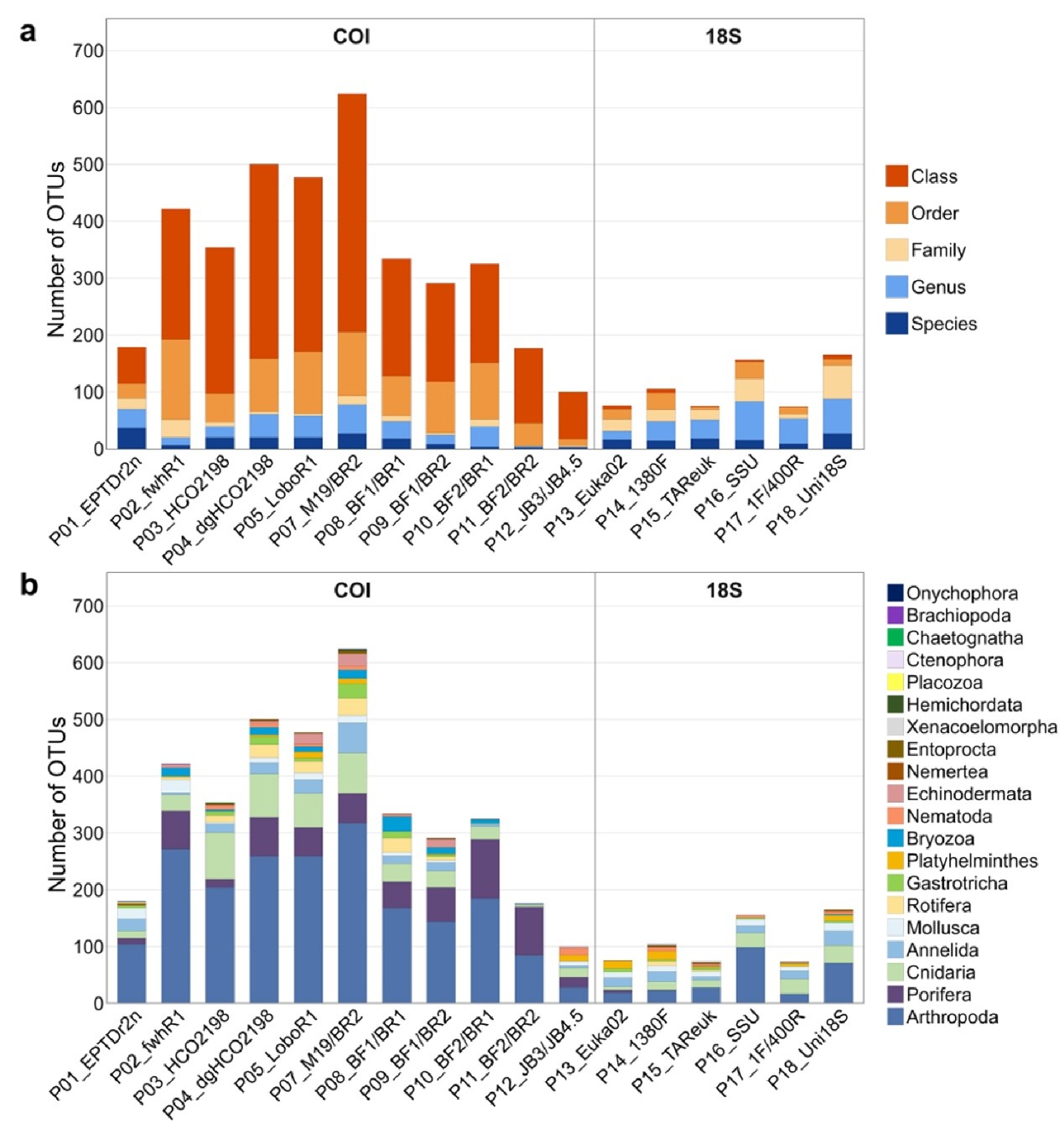
图5. 基于海洋eDNA样本的in vitro PCR分析结果,展示各引物的无脊椎分类分辨率(a)及门级类群组成(b)
昆虫是淡水中检测到最主要类群,10个OTU数量最多的目中5个均属于昆虫纲(图6)。引物P01_EPTDr2n对淡水生物指示类群EPT(蜉蝣目、襀翅目、毛翅目)昆虫的检出效果最佳,共检出165个OTU。
此外,研究还发现序列参考数据库的选择会影响分类注释结果:COI序列用BOLD数据库注释的无脊椎动物OTU数量多于GenBank,18S序列用SILVA数据库注释的结果则略优于GenBank;同时,公共数据库中无脊椎动物序列存在较大缺口,部分类群的序列数据匮乏,制约了物种的鉴定精度。

图6. 基于淡水eDNA样本in vitro分析的主要无脊椎动物目级OTU丰度热图
本研究是目前最为全面系统的无脊椎动物宏条形码引物比较研究。利用多种自然生境eDNA的分析进一步明确了不同引物在淡水与海洋环境中的适用性与局限性。研究结果为从事水生生物监测、生态评估与保护的研究人员提供了引物选择、数据库选择与实验设计的重要参考,有助于推动eDNA技术进一步标准化,提升跨区域研究数据的可比性,从而更准确、全面地评估水生态系统健康与监测生物多样性动态。
北京大学生命科学学院博雅博士后陈晓宇为本论文的第一作者,北京大学城市与环境学院博士生闫姿伶为论文的共同作者,姚蒙为论文的通讯作者。该研究得到了国家自然科学基金、国家科技基础资源调查等的支持。
© 版权声明
本文由分享者转载或发布,内容仅供学习和交流,版权归原文作者所有。如有侵权,请留言联系更正或删除。


















研究数据很扎实,对无脊椎动物多样性调查帮助很大,希望能推动标准化!
有没有可能把这些引物应用到更广泛的淡水监测项目里?🤔
COI和18S引物各有优势,看来实际应用得根据环境来选,挺有意思的。
这篇研究挺实用的,引物选择对eDNA监测确实关键。