西北农林科技大学(767)信息工程学院李富义教授课题组在蛋白质功能注释研究上取得新进展
文章导读
蛋白质功能预测的难题正困扰着生物医学界?西北农林科技大学李富义教授团队开发的ENGINE框架给出了突破性解决方案。这项发表于《Genome Biology》的研究创新性地融合等变图卷积网络与蛋白质大语言模型,首次实现三维结构与序列信息的统一整合。实验证明,该模型在预测精度和泛化能力上全面超越现有方法,更关键的是具备可解释性——能精准定位关键残基,揭示功能特异性结构特征。研究团队已开源代码并提供图形界面工具,让每位研究者都能轻松实现精准蛋白功能注释。
— 内容由好学术AI分析文章内容生成,仅供参考。
近日,信息工程学院智能计算与农业信息系统学科团队李富义教授课题组在蛋白质功能注释研究方面取得重要进展,研究成果以“A scalable equivariant graph network framework for precise protein function prediction”为题发表在《Genome Biology》。课题组硕士研究生冉子旭、博士研究生生郭旭东和澳大利亚Monash大学博士研究生潘彤为论文共同第一作者,李富义教授为通讯作者,澳大利亚Monash大学Jiangning Song教授为并列通讯作者。
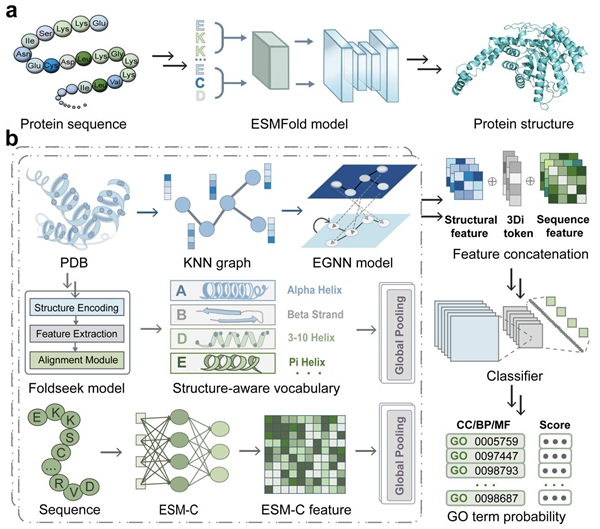
图1 ENGINE模型框架图
蛋白质功能研究有助于理解细胞内发生的复杂生物过程。然而,蛋白质结构与功能的复杂性,以及蛋白质序列数据的快速增长,使得开发高效计算方法进行精准蛋白质注释成为迫切挑战。该研究提出ENGINE多通道深度学习框架,旨在实现稳健的蛋白质功能预测。ENGINE采用等变图卷积网络捕捉蛋白质的三维结构特征,借助蛋白质大语言模型(ESM-C和Foldseek)提取进化信息和表征三维序列,实现空间结构与序列信息的统一整合。
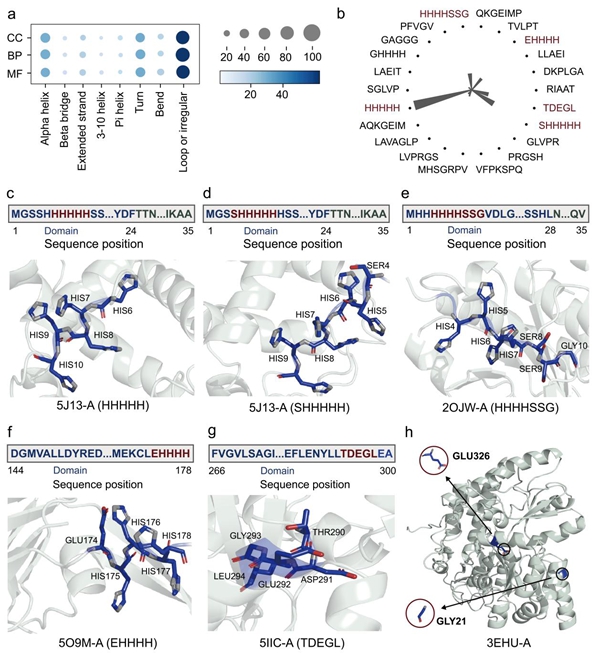
图2 ENGINE揭示了功能特异性的蛋白质二级结构特征与子结构
实验结果表明,ENGINE在蛋白质功能预测基准测试中超越现有方法,展现出强大的泛化能力与高预测精度。除性能优势外,ENGINE分析了可解释的关键结构特征,助力识别未注释的蛋白功能。同时,注意力机制的融入使ENGINE能精准定位参与多样化生物活性的关键残基,实现残基层面的功能注释。通过提供具有生物学可解释性的可靠预测,ENGINE为推进细胞过程和疾病机制的研究做出了贡献。为了便于研究人员轻松复现并拓展该工作,ENGINE已经开源,并在GitHub (https://github.com/ABILiLab/ENGINE) 和Zenodo (https://doi.org/10.5281/zenodo.17221153) 上提供配备了图形用户界面(GUI)的计算软件和源代码。此外,课题组面向校内用户提供批量计算服务。
该研究得到了国家自然科学基金[62202388]、国家重点研发计划[2022YFF1000100]和秦创原创新创业人才项目[QCYRCXM-2022–230]的支持。
编辑:张晴
终审:刘玉峰
© 版权声明
本文由分享者转载或发布,内容仅供学习和交流,版权归原文作者所有。如有侵权,请留言联系更正或删除。




















开源代码太贴心了!正好最近在写相关论文,可以借鉴一下实现思路
ENGINE框架整合了结构和序列信息,这种多模态思路确实值得学习
这个蛋白质预测模型听起来很厉害,希望能尽快应用到实际研究中👍