研究构建综合性的新生RNA数据库
文章导读
研究构建了综合性的新生RNA数据库NaRaDa,收录涵盖22个物种、415项研究的3664个新生RNA测序数据集,提供质控信息以筛选高质量数据。数据库鉴定了558574个转录调控元件,并支持在线分析转录调控动态,如表达差异、RNA聚合酶的暂停/释放转变等。旨在为解析基因转录起始变化、RNA代谢动力学及调控网络提供资源。相关成果发表于《The Innovation Life》,研究获得国家自然科学基金委员会、科技部等支持。
— 内容由好学术AI分析文章内容生成,仅供参考。
动态解析基因组转录调控是解读细胞命运和疾病发生机制的关键。然而,传统RNA测序技术受限于高丰度稳态RNA,难以捕捉转录活动的即时变化。近年来,新生RNA测序技术成为实时监测转录动态的关键工具。相较于传统RNA测序技术,新生RNA测序技术可从新生RNA即时表达情况、转录暂停情况及非编码转录调控元件的活跃情况三个层面提供转录调控信息。
近日,中国科学院广州生物医药与健康研究院研究员鲍习琛团队收集并整理了涵盖22个物种、415项研究的3664个新生RNA测序数据集,构建了综合性的新生RNA数据库NaRaDa,并提供样本质控信息,以确保用户可筛选高质量数据集。进一步,NaRaDa鉴定了558574个来自不同物种的转录调控元件,用户可通过感兴趣的基因来探索,也可通过筛选具有特定处理或实验条件的项目来获取其对应调控元件。同时,NaRaDa支持用户在线分析不同新生RNA数据集的转录调控变化,包括新生RNA表达差异、RNA聚合酶的暂停/释放转变以及转录调控元件的活跃程度变化。用户可下载并利用上述信息,深入剖析基因和调控元件在各种情况下的转录动力学。
该研究构建了综合性的新生RNA数据库,为解析转录起始变化、RNA代谢动力学及基因调控网络提供了资源库。
相关研究成果以NaRaDa: A comprehensive nascent RNA database为题,发表在The Innovation Life上。研究工作得到国家自然科学基金委员会、科学技术部等的支持。
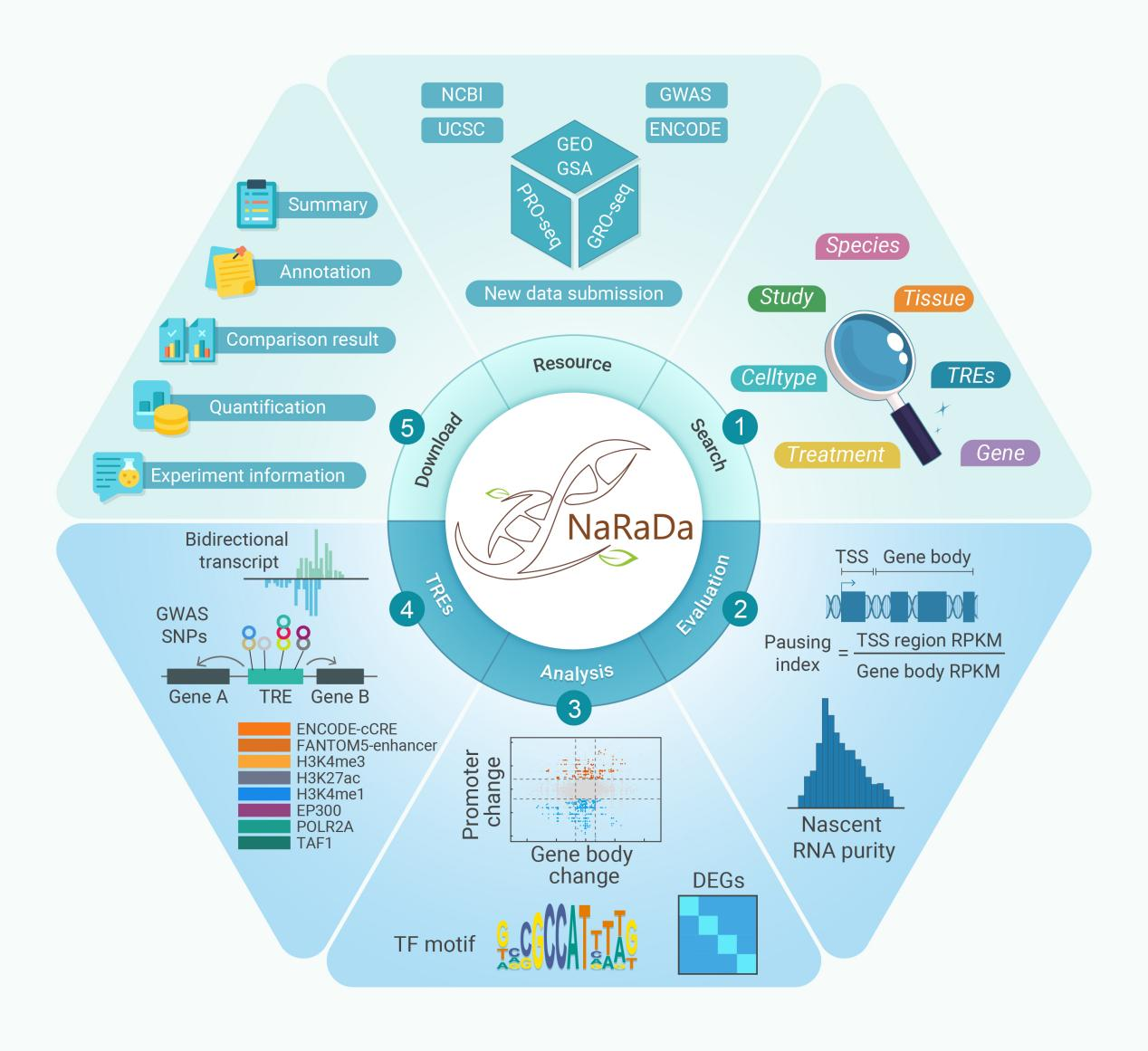
NaRaDa的整体框架
© 版权声明
本文由分享者转载或发布,内容仅供学习和交流,版权归原文作者所有。如有侵权,请留言联系更正或删除。
相关文章

暂无评论...