文章导读
你是否也被“复杂聚糖难以解析”的难题卡住?南京大学黄硕团队在Science Advances提出一种基于PBA修饰MspA纳米孔的“碎片拼接”测序平台:先用糖苷酶将聚糖切成可识别的糖片段,通过纳米孔采集信号比对自建数据库并借助分类模型(识别准确率94.1%)逐一指认碎片,最终重构出完整聚糖序列。该方法无需标记、分辨率高,并已成功测序多种天然寡糖,意味着对复杂、支链聚糖结构的高精度解析迈出重要一步。
— 内容由好学术AI分析文章内容生成,仅供参考。
聚糖在自然界中广泛存在,并且在生命活动中起着关键作用。解析聚糖复杂且分支的结构对于理解其功能至关重要。传统分析方法在解析聚糖结构(如同分异构体区分、复杂光谱解析)方面往往存在局限。
纳米孔是一种新兴的单分子传感器,广泛用于DNA和RNA的测序。纳米孔在核酸测序方面的成功激励了蛋白质和聚糖纳米孔测序的发展。但由于聚糖的结构复杂性,纳米孔聚糖测序的发展明显滞后。目前纳米孔糖分析的研究集中在少数糖的传感与区分方面,但是通用的高分辨率的纳米孔聚糖分析方法尚未被提出。
近日,南京大学黄硕教授团队基于苯硼酸(PBA)修饰的耻垢分枝杆菌膜蛋白A纳米孔(MspA-90PBA)首次提出了一种创新的“碎片拼接”纳米孔聚糖测序平台,具备高通用性、高分辨率、无标记识别的优势,能够对复杂聚糖结构进行高精度解析。首先,MspA-90PBA可以传感从单糖到聚糖以及支链糖结构,作者结合传感数据构建了纳米孔聚糖数据库(图1A)和分类模型(图1B)。分类模型对数据库中的聚糖纳米孔事件识别准确率可达94.1%。对于碎片拼接聚糖测序而言,准确识别整个聚糖包含的特征糖碎片是必要的,使用糖苷酶将聚糖水解成为糖碎片,结合对纳米孔糖数据库的搜索,聚糖的碎片组成可以被一一指认,从而可以重构出整个聚糖的序列(图1C)。使用上述方法,作者成功对棉子糖,水苏糖和毛蕊花糖进行了“碎片拼接”纳米孔测序(图1D-J)。
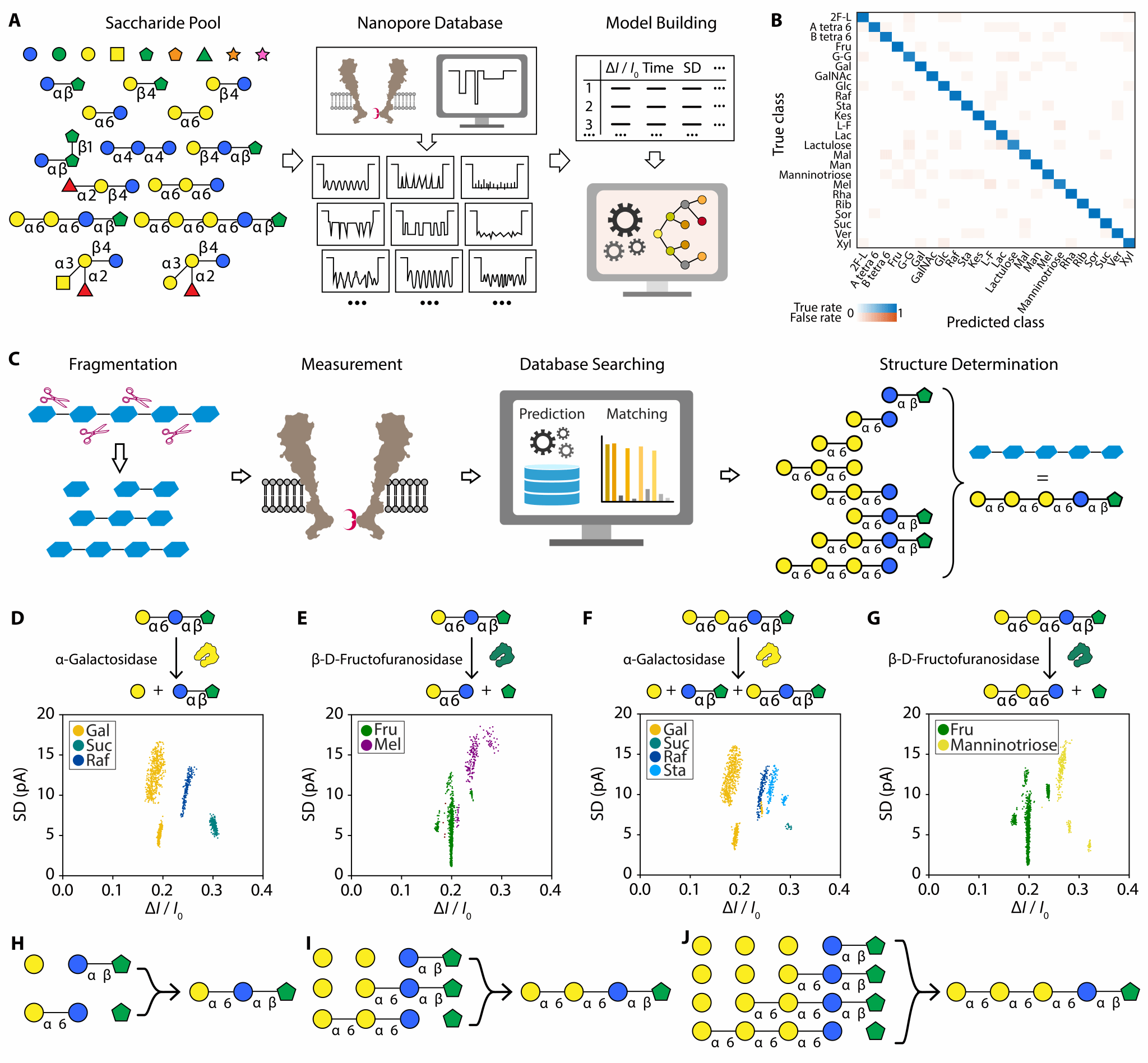
图1:基于MspA-90PBA的“碎片拼接”纳米孔聚糖测序平台的构建与应用
该工作以“Nanopore profiling and structure determination of oligosaccharides by fragmentation”为题,于2026年1月1日在《Science Advances》发表(论文链接: https://www.science.org/doi/10.1126/sciadv.aea8462)。南京大学黄硕教授为该论文的通讯作者,南京大学化学学院博士生肖云麒和已毕业博士生张善雨为该论文共同第一作者。此项研究得到了科技部国家重点研发计划、国家自然科学基金、中央高校基本科研业务费资助项目、南京大学生命科学分析化学国家重点实验室的资助,以及生命分析化学国家重点实验室和南京大学化学和生物医药创新研究院(ChemBIC)的重要支持。
© 版权声明
本文由分享者转载或发布,内容仅供学习和交流,版权归原文作者所有。如有侵权,请留言联系更正或删除。



















棉子糖和水苏糖的测序结果对比看着挺清晰的
我们实验室也在做类似研究,这个数据库构建思路值得参考
MspA-90PBA的识别准确率94.1%算高吗?有没有对比数据?
之前做糖分析总被复杂光谱搞晕,这个方法感觉实用多了
这个拼接策略挺有意思的,能解决异构体区分问题吗?