文章导读
当细菌面临"饥饿危机",如何精准调控基因表达?清华团队首次揭示T-box核糖开关的分子密码:通过"身份识别+状态感知"两步机制,动态切换构象调控翻译!研究发现,未负载氨基酸的tRNA会触发"AntiS"构象启动基因翻译,而已负载氨基酸的tRNA则诱导"查封者"构象抑制翻译。关键linker区域如同分子开关,共转录调控特性让细菌能实时响应营养变化。这项发表在《自然·通讯》的研究,为攻克结核分枝杆菌等耐药菌提供了全新靶点。
— 内容由好学术AI分析文章内容生成,仅供参考。
T-box核糖开关是一类重要的基因调控元件,广泛分布于革兰氏阳性菌中。其核心功能是通过识别tRNA 3΄末端的氨酰化状态感知细菌营养水平,进而精准调控相关基因表达,对维持细菌生长平衡至关重要。由于该元件在细菌中普遍存在而在哺乳动物中尚未发现,针对T-box核糖开关的药物设计已成为新型抗生素研发的重要方向。T-box的功能实现高度依赖其三维结构与构象动态特征。尽管科研人员已成功解析多种T-box-tRNA复合物的静态三维结构,但T-box如何精准鉴别不同氨酰化状态的tRNA,并启动后续转录或翻译调控的构象动态机制,长期以来仍是领域内亟待解决的科学问题。
11月25日,清华大学生命科学学院陈春来研究组与中国科学院生物物理研究所方显杨研究组合作,以“tRNA依赖的构象动态与折叠协调全长T-box核糖开关的翻译调控”(tRNA-dependent conformational dynamics and folding coordinate translational regulation by a full-length T-box riboswitch)为题在《自然·通讯》(Nature Communications)在线发表研究论文。研究首次运用单分子荧光共振能量转移(smFRET)技术,系统探究了结核分枝杆菌(Mycobacterium tuberculosis)ileS T-box核糖开关的结构动态,揭示了其在共转录水平鉴别tRNA 3΄末端氨酰化状态、调控翻译起始过程的分子机制,为深入理解RNA-RNA相互作用的功能机制及新型抗生素开发提供了重要理论支撑。
应用smFRET技术开展RNA构象动态研究的核心前提是实现RNA的高效位点特异性荧光标记。受传统标记技术限制,以往基于smFRET的RNA动态研究多局限于短链RNA。研究团队基于NaM-TPT3非天然碱基(UBP)系统,近期成功开发长链RNA位点特异性标记技术,这一技术突破为全长T-box(200-300核苷酸)的构象动态研究奠定了基础。
研究团队在T-box核糖开关的适配体与鉴别结构域筛选多对标记位点,结合自主开发的RNA定点荧光标记技术与smFRET技术,系统观测了不同氨酰化状态tRNA结合诱导的T-box结构转变。结果揭示,T-box通过“构象选择”机制实现对tRNA的两步精准识别:第一步为“身份识别”——tRNA反密码子区与T-box解码结构域结合,促使Stem I/II形成稳定“对接构象”,同时触发鉴别结构域折叠,为后续氨酰化状态感知做好准备;第二步为“氨酰化状态感知”——若tRNA未负载氨基酸,其3΄末端与鉴别结构域稳定结合,使开关维持“AntiS”构象,暴露SD序列并启动下游基因翻译。
若tRNA已负载氨基酸,则诱导鉴别结构域中Stem III与linker区域呈现高度柔性,促使构象转变为“Sequestrator”(查封者),隐藏SD序列以抑制翻译。进一步突变实验证实,连接适配体与鉴别结构域的linker区域是感知tRNA氨基酸状态的关键结构单元,关键碱基突变会导致T-box完全丧失对tRNA氨酰化状态的响应能力。考虑到细菌内RNA折叠伴随转录同步进行,研究团队进一步探讨共转录过程对T-box调控机制的影响。通过制备不同长度的T-box截短体(模拟不同转录中间产物)及荧光标记的共转录T-box/tRNA复合物,并开展smFRET实验,结果表明T-box对tRNA NCCA末端的识别具有显著的转录依赖性。这一“共转录调控”特性使T-box能够快速响应细胞内氨基酸水平变化,实现基因表达的高效精准调控。
基于上述系列发现,研究团队提出T-box核糖开关的共转录调控模型(图1),首次系统阐明了其在转录过程中分步识别tRNA、鉴别氨酰化状态、动态切换构象并最终调控基因表达的完整动态过程。
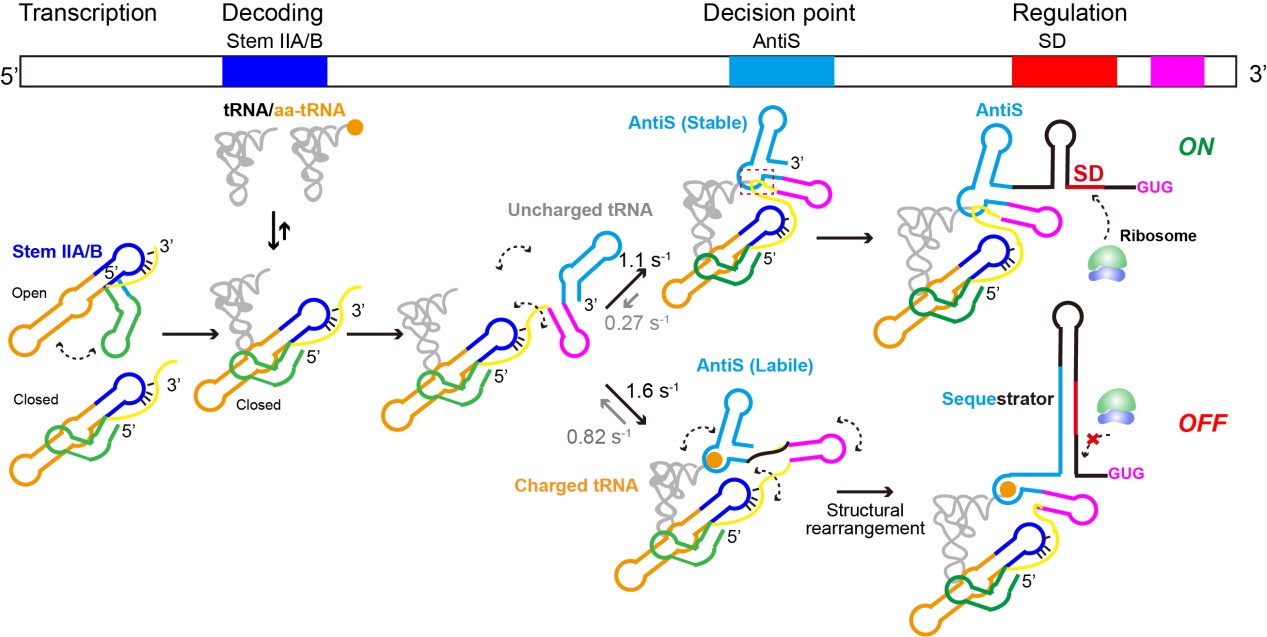
T-box核糖开关在共转录水平上调控基因表达的模型
中国科学院生物物理研究所研究员方显杨、清华大学生命科学学院副教授陈春来为论文共同通讯作者。清华大学已出站博士后牛晓林、新疆大学2021级联培博士生蔡珊珊为论文共同第一作者。
研究得到国家重点研发计划、国家自然科学基金委、北京市生物结构前沿研究中心、中国博士后科学基金以及清华大学“水木学者”计划等的支持。
论文链接:
https://doi.org/10.1038/s41467-025-65388-6
供稿:生命学院
编辑:李华山
审核:郭玲
© 版权声明
本文由分享者转载或发布,内容仅供学习和交流,版权归原文作者所有。如有侵权,请留言联系更正或删除。




















清华和生物物理所的合作真是强强联合啊
希望这个发现能早日应用到临床治疗中👍
单分子荧光共振能量转移技术原来这么厉害🤔
这项研究对新型抗生素研发很有意义!